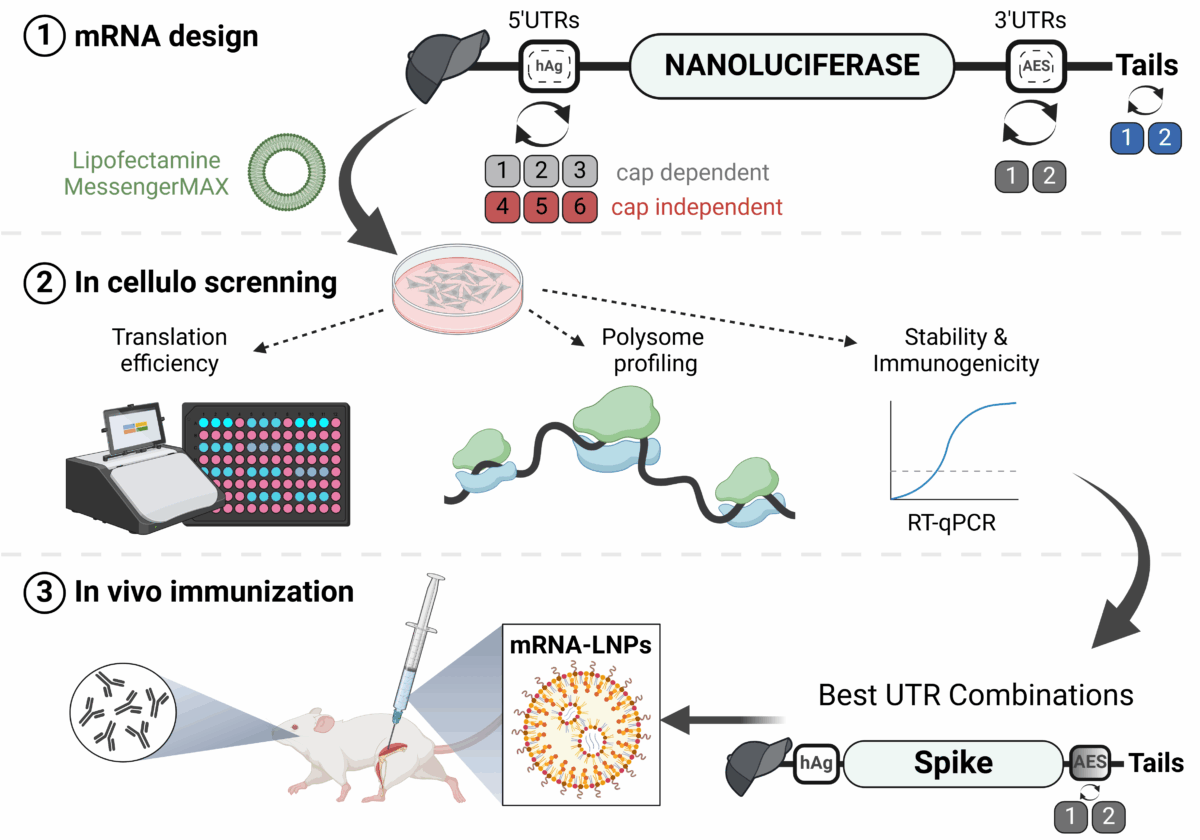
L'ARN messager (ARNm) s’est imposé comme une nouvelle technologie prometteuse dans le domaine des médicaments. L’efficacité de la technologie ARNm dépend à la fois de l'efficacité de la délivrance de l'ARNm et de sa traduction. Les régions non traduites (UTR) et la queue poly(A) jouent un rôle crucial dans la régulation de la cinétique intracellulaire de l’ARNm. Dans le but d'améliorer le potentiel thérapeutique de l’ARNm synthétique, nous avons évalué différentes UTRs et queues, en utilisant les séquences du vaccin contre la COVID-19 de Pfizer-BioNTech comme référence.
Les chercheurs du CBM ont d’abord testé six 5’ UTRs (dépendantes/indépendantes de la coiffe), évalué neuf combinaisons de 5’ UTRs et 3’ UTRs, ainsi qu’une nouvelle queue hétérologue A/G dans des modèles cellulaires et in vivo en utilisant la luciférase comme gène rapporteur. Ensuite, pour décrypter le mécanisme de traduction des UTRs sélectionnées, ils ont corrélé l’expression de l’ARNm avec l’interaction avec les ribosomes, la demi-vie des ARNm, leur immunogénicité et la structure des UTRs.
Leurs résultats ont montré que la queue hétérologue qu'ils ont introduite est aussi puissante que celle utilisée par Pfizer-BioNTech, et ils ont confirmé la forte efficacité de la 5’ UTR de l’alpha-globine humaine. Ils ont également révélé le potentiel des 3’ UTRs de VP6 et SOD. Ils ont validé leurs résultats en utilisant un ARNm codant pour la protéine Spike du SARS-CoV-2 formulé en nanoparticules lipidiques (LNP) pour l’immunisation de souris. Dans l’ensemble, les 3’ UTRs sélectionnées et la queue hétérologue A/G présentent un fort potentiel en tant que nouveaux éléments pour la conception d’ARNm thérapeutiques.
Ces résultats ouvrent de nouvelles perspectives pour les thérapies à ARNm. Si des améliorations restent à apporter pour atteindre une expression supérieure aux stratégies existantes, cette stratégie contribue à améliorer les thérapies ARNm.
Ces résultats sont liés à un brevet
Référence :
Evaluation of synthetic mRNA with selected UTR sequences and alternative Poly(A) tail, in vitro and in vivo. Medjmedj A, Genon H, Hezili D, Ngalle Loth A, Clemençon R , Guimpied C, Mollet L, Bigot A, Wien F, Hamacek J, Chapat C, Perche F, Molecular Therapy Nucleic Acids 2025.