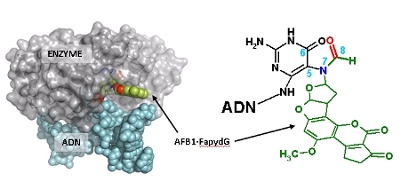
Le défi pour les ADN glycosylases telles que Fpg et hOgg1, enzymes qui initient le système de réparation par excision de base, est de trouver et d’éliminer une base endommagée parmi un million de bases normales dans l’ADN. Ce processus est d’autant plus complexe que ces enzymes doivent identifier des lésions de petites tailles qui ne sont pas associées à des changements importants de la structure locale de l’ADN mais aussi, des lésions encombrantes qui déforment fortement la structure de l’ADN, tout cela, en utilisant le même site actif. En combinant la synthèse chimique de molécules d’ADN contenant des modèles de lésions variant par leur encombrement stérique et une analyse structurale par cristallographie de complexes Fpg/ADN, des chercheurs du CBM ont établi les bases moléculaires de la formation de complexes abortifs mais stables entre ce type d’enzymes et la lésion encombrante. Ils proposent que la formation de ce type de complexe moléculaire inhibe les processus de réparation des lésions encombrantes de l’ADN, expliquant ainsi la persistance de ce type de dommages observés in vivo et les effets génonotoxiques qui leur sont associés. L’inhibition sélective de Fpg et hOgg1 par des mimes synthétiques de lésions encombrantes ouvre la voie à la possibilité de moduler la réparation de l’ADN in vivo. Le ciblage pharmacologique de ce type d’enzymes pourrait être proposé dans le traitement de certains cancers (poumon et colon) et de maladies dégénératives du système nerveux central (Alzheimer et Huntington).
Référence :
Coste, F., Ober, M., Le Bihan, Y.-V., Izquierdo, M.A., Hervouet, N., Carell, T. and Castaing, B. “Bacterial base excision repair enzyme Fpg recognizes bulky N7-substituted-FapydG lesion via unproductive binding mode.” Chemistry & Biology (2008) 15, 706-717
Illustration: