[Ezzine S., Vassaux G., Pitard B., Barteau B., Malinge J.-M., Midoux P., Pichon C. and Baril P.
RILES, a novel method for temporal analysis of the in vivo regulation of miRNA expression
Nucleic Acids Research, 2013, 1-14->http://nar.oxfordjournals.org/content/early/2013/09/05/nar.gkt797.long]
Résumé :
L’interférence à ARN (RNAi) est un mécanisme ancestral de régulation post-transcriptionnelle des gènes, faisant intervenir des petits ARN non-codant (microARN). Leur hybridation avec un ARN messager cible conduit à l’arrêt de la traduction ou à la dégradation de l’ARN messager. Depuis leur découverte dans les années 80, un nombre croissant d’études a permis d’établir le rôle fondamental et essentiel des microARNs dans des processus biologiques aussi essentiels que le développement embryonnaire, l’hémostasie, le cycle cellulaire et l’apoptose. De par leur mécanisme de régulation génique séquences-spécifiques, les microARNs suscitent d’énormes intérêts pour le traitement de nombreuses pathologies tel que le cancer par exemple. La connaissance de leur mode de régulation et de leur profil d’expression est fondamentale pour leur utilisation comme agents thérapeutiques. Pour cela, un nouveau système d’imagerie moléculaire appelé RILES a été développé dans l’équipe « Transfert d’acide nucléiques par des systèmes non viraux » du CBM en collaboration avec nos partenaires Niçois et Nantais. Ce travail vient de faire l’objet d’une publication dans Nucleic Acids Research. Ce système repose sur l’ingénierie d’un vecteur d’expression inductible de façon à ce que le microARN étudié contrôle directement l’expression d’un répresseur transcriptionnel. De cette façon, la configuration du RILES est placée en mode ON (« switch-on ») permettant l’émission de signaux optiques facilement quantifiable par imagerie bioluminescente in vitro et in vivo. Dans cet article, nous décrivons la preuve de fonctionnalité du RILES in vivo à travers deux cibles : le foie et le muscle squelettique. Nos résultats montrent que RILES permet de collecter des informations pertinentes qui renseignent de façon précise sur la dynamique de régulation des microARNs au cours du développement d’un processus physiopathologique. L’exploitation de RILES pourrait avoir des retombées dans les approches de médecine personnalisée de demain qui consisteront à traiter chaque patient de façon individuelle en tenant compte du profil d’expression génique des cibles thérapeutiques.
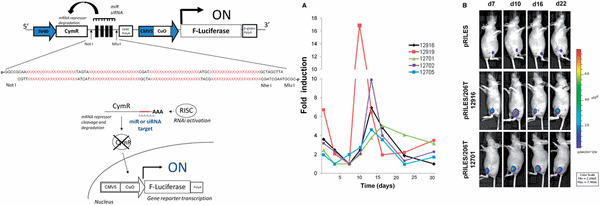
Figure de gauche :
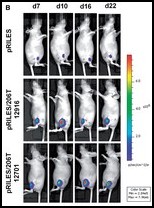
Schematic representation of the RILES method. When present in cells, target miRNA or siRNA binds to the four complementary-block sequences located in the 30UTR of the CymR repressor transcript and activates the RNAi silencing complex (RISC) machinery. The CymR mRNA is then cleaved and degraded, resulting in lack of repressor production. The luciferase expression system is thus switched-ON, generating a positive bioluminescence signal.
Figure de droite :
Real time monitoring of miRNA-206 regulation during development of skeletal muscle atrophy. Two micrograms of pRILES/206T combined with 6 mg of the pQE30 empty expression plasmid were formulated with the amphiphilic block polymer 704 and ntramuscularly injected in the tibialis anterior to transfect the skeletal muscles of nude mice. Three days later (day 0), bioluminescence activity in the lower legs of mice was measured, and the left sciatic nerves were cut surgically to induce denervation and atrophy. Mice (n=5) were thereafter scanned twice a week for the first 3 weeks and then once a week till day 35 (end point of our experiment). (A) Kinetic of miRNA-206 expression detected in each individual mouse by bioluminescence imaging. Results are expressed as relative fold of luciferase induction by normalizing the bioluminescence values to the minimal value found before the bioluminescence peak for each single mouse. (B) Representative bioluminescence images collected from one representative mouse from the pRILES group and two representative mice from the pRILES/206T group at days 7 (d7), 13 (d13), 22 (d22) and 30 (d30). The number of each mouse for identification during the longitudinal study is given.
