Cette technique permet l’analyse de mélanges de peptides provenant d’une protéolyse de protéines en solution ou en gel d’électrophorèse mono ou bidimensionnelle, ou d’autre techniques de séparation. Le spectromètre de masse est utilisé en couplage avec une HPLC spécialisée pour les très faibles débits (nL/min), et en mode tandem (MS/MS) pour fragmenter les ions.
La fragmentation des peptides permet d’identifier la protéine dont ils sont issus. Les centaines de spectres de fragmentation obtenus lors d’un même run nanoLC-MS/MS permettent d’identifier simultanément plusieurs protéines. Cette approche peut également donner la localisation précise des modifications post-traductionnelles (phosphorylation par exemple) ou d’autres modifications chimiques sur les peptides.
La trappe ionique est utilisée en infusion directe, sans être couplée à la chromatographie, dans le cadre de projets de recherche (par exemple, analyse de complexes protéine/ligand).
PRINCIPE DE FONCTIONNEMENT
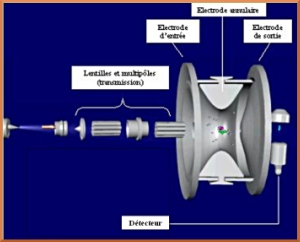
Cet analyseur comporte une électrode annulaire et deux calottes sphériques. A ces trois éléments sont appliquées des tensions, f0 pour les calottes et -f0 pour l’électrode annulaire. Ces valeurs de tensions permettent de garder les ions formés captifs au sein de la trappe. Ils y suivent une trajectoire en forme de huit en trois dimensions.
En fonction de leur rapport m/z, les ions vont être expulsés de la trappe vers le détecteur.
AVANTAGES DE LA TRAPPE IONIQUE
- Possibilité de faire de la fragmentation d’ions
- Piégeage d’ions se traduit par une bonne sensibilité
- Une adaptabilité à différentes techniques de couplage comme dans le cas de la nanoHPLC-MS
