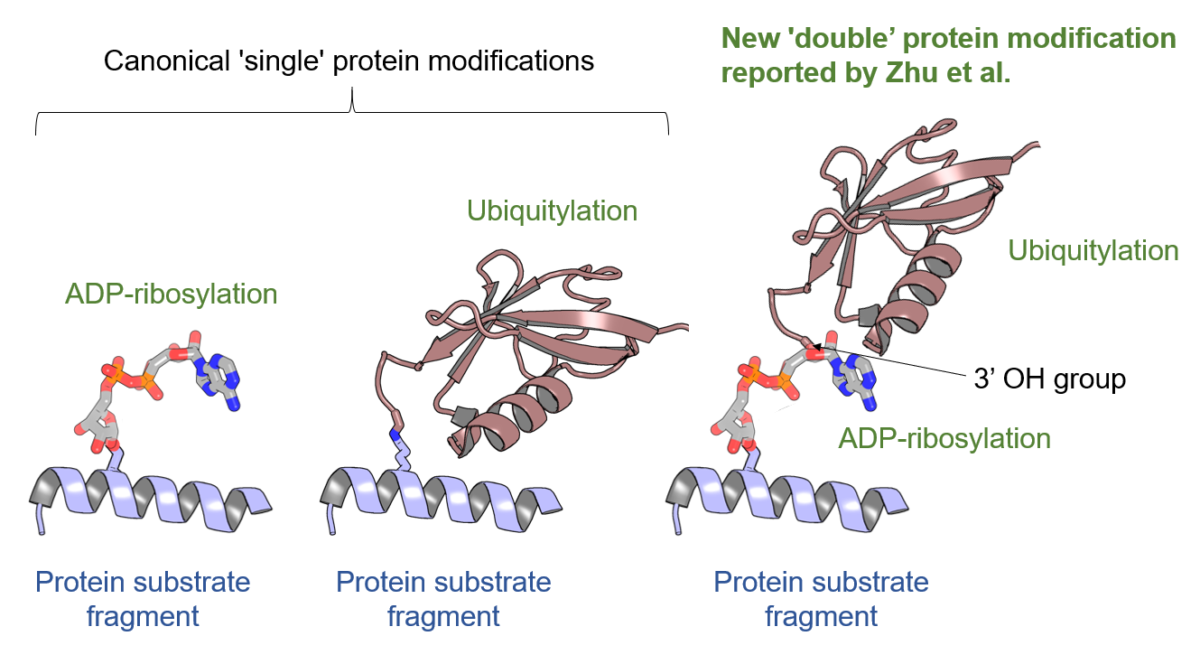
Proteins are the main ‘molecular machines’ of the cell. To efficiently perform their tasks, they have to be dynamically switched on and off, recruited to specific cellular locations, and degraded in a timely manner. One of the main mechanisms that regulate these processes is temporary covalent attachment, to a protein, of extra regulatory elements known as protein post-translational modifications. The modification reaction is catalysed by specific enzymes and can lead to changes in protein activity, localisation, or half-life. Two of the common protein modifications are ubiquitin and ADP-ribose, each of which can be linked directly to a protein substrate.
In the study published in Science Advances, an international team of researchers, including Vincent Aucagne, Marcin Suskiewicz, and Hervé Meudal from the CBM in Orléans, led by Ivan Ahel and Dragana Ahel groups at the University of Oxford, have demonstrated that these two individual modifications can be joined together, producing a ‘double’ protein modification. The enzymes responsible for this process are DELTEX E3 ligases, which can efficiently attach ubiquitin to protein-linked ADP-ribose. A key contribution of Orléans scientists to the project was the analysis of the ubiquitin-ADP-ribose linkage performed using mass spectrometry (MS) and nuclear magnetic resonance (NMR) equipment of the new MOV2ING platform in Orléans.
The study shows that different protein modifications can be joined together to either combine two regulatory signals or produce a third, distinct signal, with a specific function. This shows previously unappreciated level of complexity in protein regulation.
While the role of ubiquitin-ADP-ribose in cells remains unclear, DELTEX enzymes have previously been linked to both development and antiviral response. The authors showed that the SARS-CoV-2 virus possesses enzymes that can remove the new modification, possibly allowing the virus to inhibit the host immune response.
Références :
Kang Zhu, Marcin J. Suskiewicz, Hloušek-Kasun, Hervé Meudal, Andreja Mikoč, Vincent Aucagne, Dragana Ahel and Ivan Ahel
DELTEX E3 ligases ubiquitylate ADP-ribosyl modification on protein substrates
Science Advances, 5 Oct 2022, Vol 8, Issue 40 DOI: 10.1126/sciadv.add4253