Bien que chaque cellule de notre corps contienne la même information génétique, les cellules diffèrent dans la manière dont elles l'utilisent, un processus connu sous le nom "d'expression génétique". La régulation de l'expression des gènes est orchestrée par des protéines appelées facteurs de transcription, qui se lient à des séquences spécifiques de l'ADN. Les facteurs de transcription sont traditionnellement considérés comme opérant principalement sous forme de molécules uniques ou de dimères.
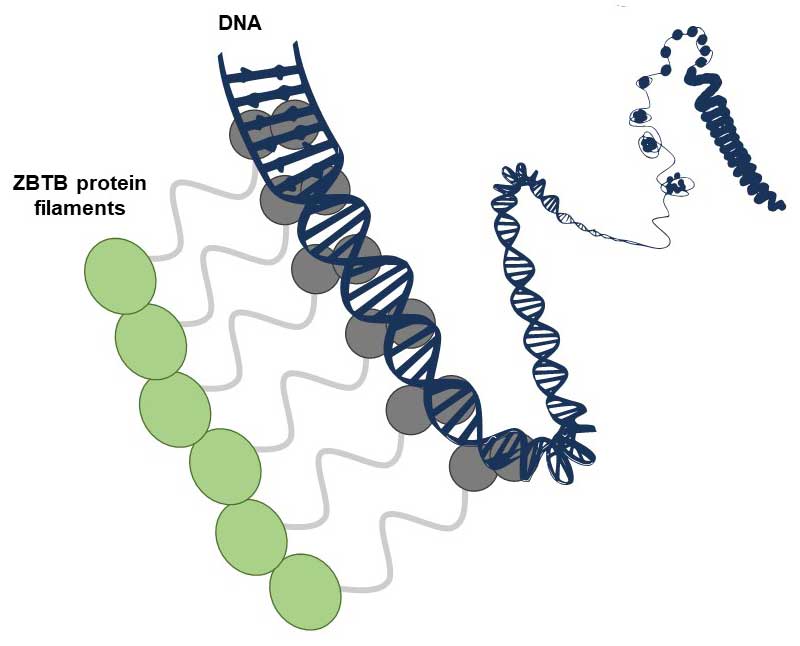
L'article de Mance et al. révèle que plusieurs facteurs de transcription de la famille connue sous le nom de ZBTB, présents chez l'homme et d'autres animaux, ont la capacité de former des structures filamenteuses non covalentes composées de nombreuses copies identiques de protéines disposées en chaîne. Au niveau moléculaire, de telles structures pourraient offrir des avantages significatifs pour se lier à l'ADN, qui est lui-même une molécule allongée contenant de nombreuses séquences répétées. Quelques exemples de facteurs de transcription formant des filaments avaient déjà été rapportés, mais cette étude étend ce concept à une grande famille de cette protéine aux fonctions importantes. L'étude - qui combine des analyses structurales, biophysiques et fonctionnelles réalisées in vitro et dans des cellules - a été réalisée par l'équipe "Modifications post-traductionnelles et réparation de l'ADN" du CBM et leurs collaborateurs à Orléans, Rennes et Marseille, dont l'équipe "Spectrométrie de masse fonctionnelle des assemblages moléculaires" également au CBM.
Les résultats de cette recherche, ainsi qu'une étude complémentaire réalisée par les groupes de Benjamin Ebert et Eric Fischer du Dana-Farber Cancer Institute à Harvard (publiée dans le même numéro de Molecular Cell), remettent en question la vision traditionnelle de la fonctionnalité des facteurs de transcription.
Dans les cellules, les protéines ZBTB sont régulées par un processus appelé SUMOylation, qui consiste à leur ajouter une petite étiquette appelée SUMO, ce qui modifie leur fonctionnement. Les études sur les protéines ZBTB entreprises à Orléans, au cours desquelles les structures filamenteuses ont été découvertes, font partie du projet "SUMOwriteNread" financé par l'Union européenne (subvention ERC n° 101078837). Les chercheurs étudient actuellement l'interaction entre la capacité à former des filaments et le marquage SUMO pour comprendre la réalité complexe de la régulation de l'expression des gènes.
Cette recherche a été signalée par CNRS Chimie sur son site internet.
Dynamic BTB-domain filaments promote clustering of ZBTB proteins.
Lucija Mance, Nicolas Bigot, Edison Zhamungui Sánchez, Franck Coste, Natalia Martín-González, Siham Zentout, Marin Biliškov, Zofia Pukało, Aanchal Mishra, Catherine Chapuis, Ana-Andreea Arteni, Axelle Lateur, Stéphane Goffinont, Virginie Gaudon, Ibtissam Talhaoui, Ignacio Casuso, Martine Beaufour, Norbert Garnier, Franck Artzner, Martine Cadene, Sébastien Huet, Bertrand Castaing & Marcin Józef Suskiewicz
Molecular Cell 2024
https://doi.org/10.1016/j.molcel.2024.05.029