Nos cellules sont constamment exposées aux espèces réactives de l’oxygène qui peuvent induire des lésions sur l’ADN et compromettre ainsi la stabilité des génomes nucléaire et mitochondrial et sont à l’origine de plusieurs maladies humaines telles que le cancer ou les maladies neurodégénératives.
L'ADN glycosylase OGG1 supervise la détection et l'élimination de la 7,8-dihydro-8-oxoguanine (8-oxoG), qui est une des bases oxydées la plus fréquemment induite par le stress oxydant. Cette lésion très mutagène a peu d’effet sur la structure native de l’ADN et sa détection nécessite une inspection minutieuse des bases par OGG1 via un mécanisme qui n'est que partiellement compris.
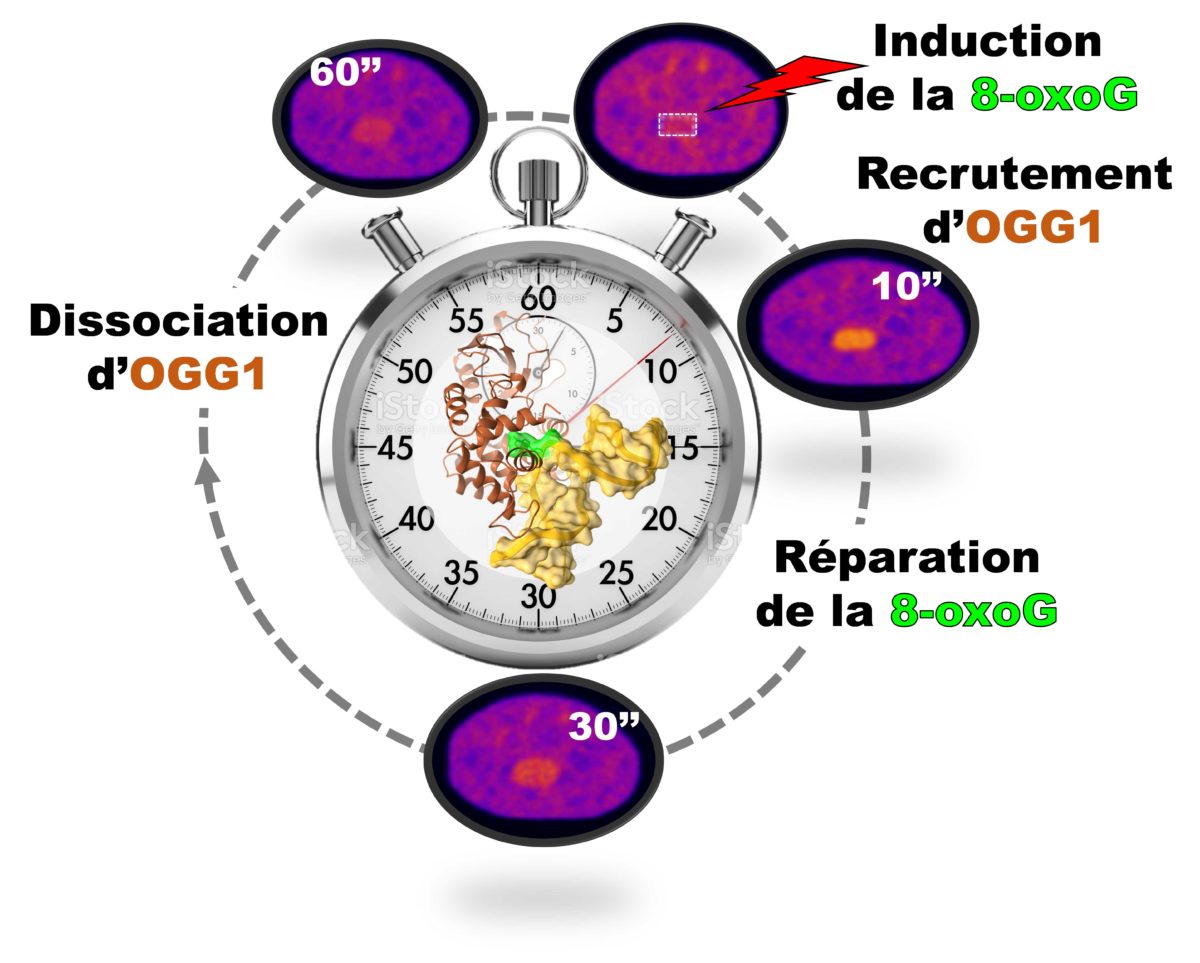
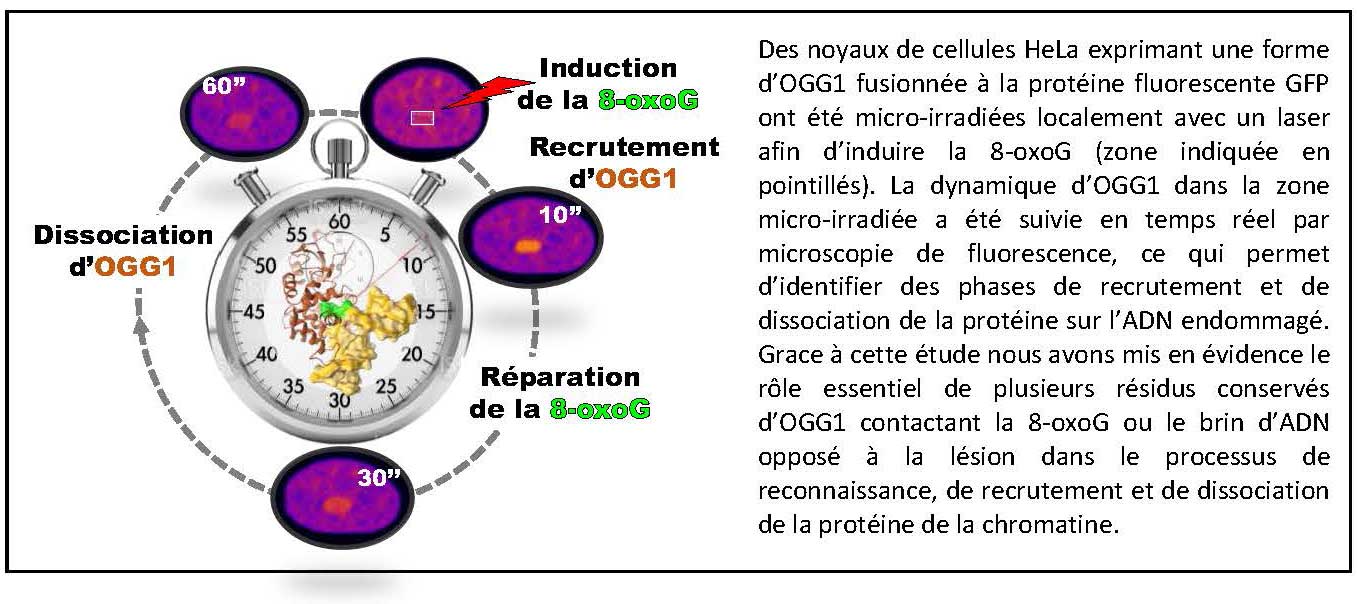
Des chercheurs du CBM, en collaboration avec les équipes de Sébastien Huet (IGDR, Université de Rennes) et d’Anna Campalans (IRCM, CEA-Fontenay-aux-Roses), ont caractérisé ce processus grâce à une complémentarité d’approches allant de l’analyse biochimique de l’interaction entre OGG1 et l’ADN à l’imagerie de cellules vivantes exprimant OGG1 marqué par fluorescence. Les données obtenues mettent en évidence le rôle essentiel de résidus conservés d’OGG1 contactant la 8-oxoG ou le brin d’ADN adjacent, dans le processus de détection et d’accumulation des lésions. La dynamique d'OGG1 a été suivie dans le noyau de cellules humaines en situation basale et après l’induction de la 8-oxoG par micro-irradiation laser. Il a été observé que OGG1 échantillonne constamment l'ADN en alternant rapidement entre la diffusion dans le nucléoplasme et de courts transits sur l'ADN. Le processus d'échantillonnage est étroitement régulé par le résidu conservé G245 qui est crucial pour le recrutement rapide d'OGG1 sur les lésions oxydatives induites par micro-irradiation laser. De plus, les résidus Y203, N149 et N150, impliqués dans les premiers stades de la recherche de la 8-oxoG, régulent de façon différentielle l’identification de la lésion et le recrutement de la protéine de réparation sur les lésions oxydatives.
Ostiane D’Augustin, Virginie Gaudon, Capucine Siberchicot, Rebecca Smith, Catherine Chapuis, Jordane Depagne, Xavier Veaute, Didier Busso, Anne-Marie Di Guilmi, Bertrand Castaing, J Pablo Radicella, Anna Campalans, Sébastien Huet
Identification of key residues of the DNA glycosylase OGG1 controlling efficient DNA sampling and recruitment to oxidized bases in living cells
Nucleic Acids Research, 2023;, gkad243, https://doi.org/10.1093/nar/gkad243
Contact : bertrand.castaing@cnrs-orleans.fr