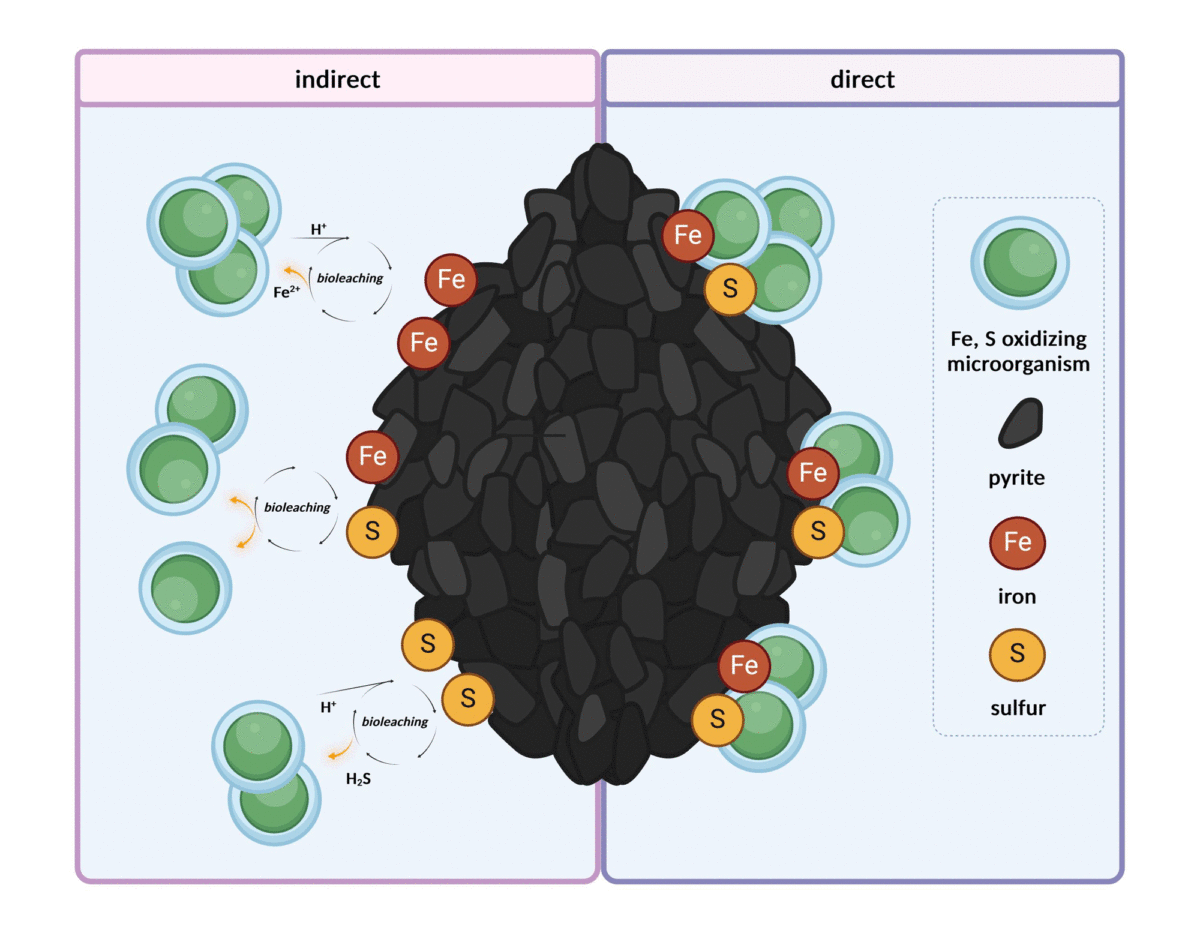
Nous avons étudié le métabolome de l'archéon Metallosphaera sedula, extrêmement thermoacidophile et oxydant le fer et le soufre, cultivé sur de la pyrite minérale (FeS2). L'extraction des matières organiques de ces micro-organismes est un défi majeur en raison du contact étroit et de l'interaction entre les cellules et les matières minérales. Par conséquent, nous avons appliqué un protocole amélioré pour briser les cellules microbiennes et séparer leurs constituants organiques de la surface minérale, pour extraire les composés lipophiles par extraction liquide-liquide, et nous avons effectué des analyses métabolomiques en utilisant MALDI-TOF MS et UHPLC-UHR-Q/TOF. Grâce à cette approche, nous avons identifié plusieurs molécules impliquées dans le métabolisme central du carbone et dans la voie d'Entner-Doudoroff modifiée que l'on trouve chez les Archaea, des composés liés au métabolisme du soufre et des molécules impliquées dans l'adaptation de M. sedula à des environnements extrêmes, telles que la tolérance aux métaux et la résistance à l'acide. En outre, nous avons identifié des molécules impliquées dans les interactions microbiennes, c'est-à-dire les interactions à la surface des cellules par la formation de biofilms et les interactions cellule-cellule par le "quorum sensing", qui s'appuie sur des molécules messagères pour la communication microbienne. En outre, nous avons réussi à extraire et à identifier différentes quinones saturées contenant du thiophène à l'aide d'un logiciel d'identification avancée des composés (MetaboScape). Ces quinones sont des transporteurs d'électrons de la chaîne respiratoire chez M. sedula, avec un potentiel de biomarqueur pour la détection de la vie dans des conditions environnementales extrêmes.
Référence :
Gfellner SV, Colas C, Gabant G, Groninga J, Cadene M, Milojevic T. Improved protocol for metabolite extraction and identification of respiratory quinones in extremophilic Archaea grown on mineral materials. Front Microbiol. 2025 Jan 8;15:1473270. doi: 10.3389/fmicb.2024.1473270