Lire le communiqué de presse du CNRS
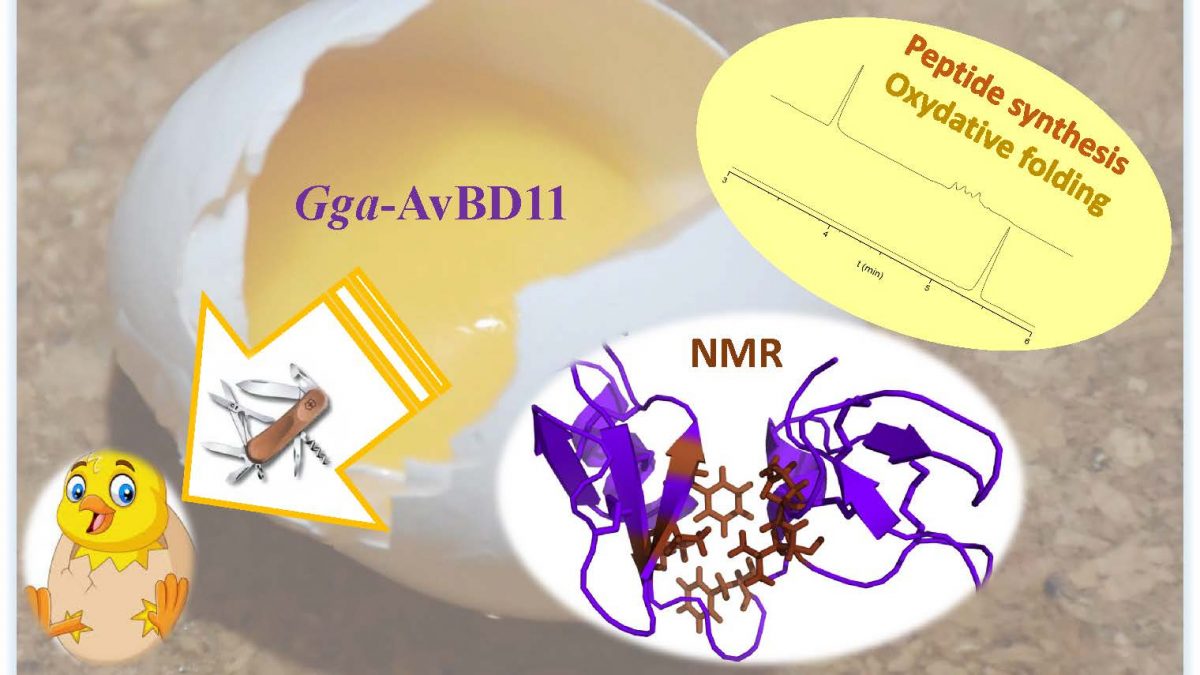
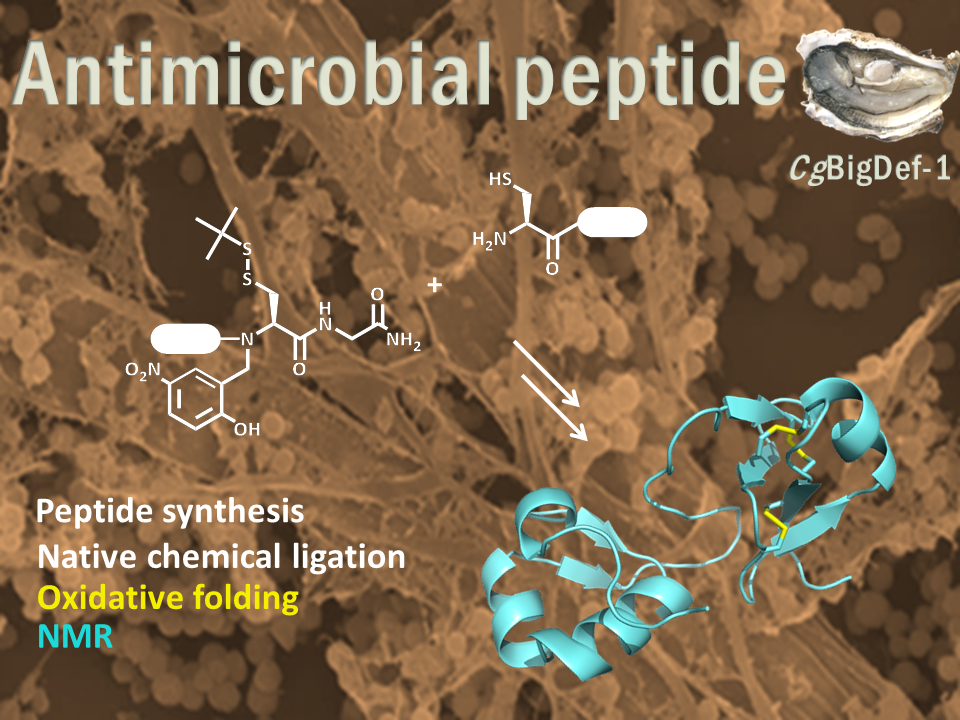
Gga-AvBD11, la β-défensine aviaire 11 du poulet commun Gallus gallus (Gga-AvBD11), est spécifique à l'œuf et représente la seule défensine de double taille (9,3 kDa) parmi les 14 AvBDs identifiées chez les espèces aviaires. L'apparition d'une telle protéine à double domaine au cours de l'évolution pourrait être motivée par une augmentation de son efficacité biologique par rapport à une molécule à simple domaine et / ou par la nécessité d'acquérir de nouvelles fonctions portées uniquement par la protéine entière. Pour évaluer la contribution des deux domaines, nous les avons synthétisés chimiquement. Nous avons résolu par RMN la structure 3D de chaque domaine, ainsi que celle de la protéine entière, composée de deux domaines β-défensine compactés. Il n'y a aucune référence pour une telle double-β-défensine dans les banques de données structurales. Ainsi, AvBD11 est l'archétype d'une nouvelle famille structurale, que nous avons nommée Av-DBD (avian-double-beta-defensin).
Sa forte conservation de séquence chez les oiseaux suggère des rôles essentiels dans l'oeuf. En collaboration avec plusieurs équipes (Nouzilly et Tours, France), nous avons montré que la protéine Gga-AvBD11 présente des activités antimicrobiennes contre les pathogènes Gram + et Gram-bactériens, le protozoaire aviaire Eimeria tenella et le virus de la grippe aviaire (H1N1). Nous avons également montré des activités cytotoxiques et anti-invasives, suggérant qu'il pourrait être impliqué dans la (re)modélisation des tissus embryonnaires. Nos résultats indiquent une importance critique du domaine cationique N-ter dans la médiation des activités antibactériennes, antiparasitaires et anti-invasives, le domaine C-ter potentialisant les deux dernières activités. De façon frappante, l'activité antivirale dans les cellules de poulet infectées nécessite la protéine entière.
L'avantage pour les espèces aviaires de posséder une double défensine est une question fascinante. Nous poursuivons les études de relations structure-activité et phylogénétiques au sein de la famille AvBD11 grâce au soutien de la Région Centre Val de Loire (projet SAPhyR-11).
Ce travail a été financé par les subventions de projets MUSE (Subvention n ° 2014-00094512) et SAPhyR-11 (Subvention n ° 2017-119983) de la Région Centre-Val de Loire.
Structure, function, and evolution of Gga-AvBD11, the archetype of the structural avian-double-β-defensin family
Nicolas Guyot, Hervé Meudal, Sascha Trapp, Sophie Iochmann, Anne Silvestre, Guillaume Jousset, Valérie Labas, Pascale Reverdiau, Karine Loth, Virginie Hervé, Vincent Aucagne, Agnès F. Delmas, Sophie Rehault-Godbert, and Céline Landon
PNAS January 7, 2020 117 (1) 337-345; first published December 23, 2019 https://doi.org/10.1073/pnas.1912941117