Les recherches de l’équipe contribuent au développement de nouveaux composés à activités antimicrobiennes ou insecticides, à l’identification de nouvelles cibles thérapeutiques, ou participe au développement de nouveaux médicaments photo-activables. Nos activités s’appuient majoritairement sur l’utilisation de la RMN en biologie structurale et en métabolomique.
Les peptides riches en ponts disulfure, peptides antimicrobiens (I) et toxines (II) sont nos sujets d’études privilégiés, et la détermination de leur structure 3D l’une des étapes dans la compréhension de leurs mécanismes d’action. Dans le contexte CoViD des dernières années, nous nous intéresserons aussi à l’analyse des structures protéiques spike pour le développement de vaccins (IV).
La métabolomique (III) quant à elle est essentiellement développée sur des modèles drosophiles de pathologies humaines, pour comprendre quelles sont les voies métaboliques impactées par les maladies.
I. PEPTIDES ANTIMICROBIENS (riches en ponts disulfure)
Les peptides antimicrobiens (PAMs), molécules clé de l’immunité innée de tous les organismes vivants, représentent aujourd’hui une des alternatives prometteuses pour lutter contre l’antibiorésistance, problème majeur de de santé publique. Nous travaillons à la compréhension fine des structures (diversités structurales, relations structures-activités, évolution des structures via la phylogénie) et la compréhension fine des mécanismes d’action de certains PAMs originaux prometteurs, pour participer à la conception des nouveaux composés antimicrobiens de demain.
Les doubles défensines d’oiseaux
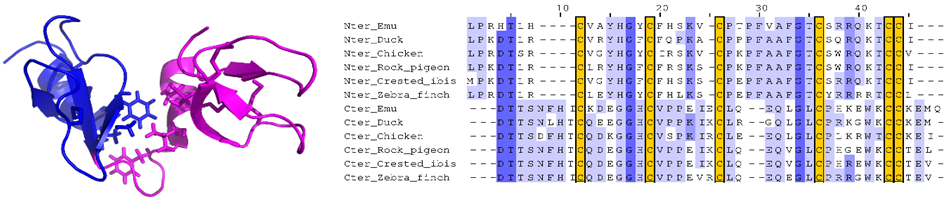
Fig1 : Nous avons étudié la structure 3D de la première double défensine aviaire, Gga-AvBD1, du poulet Gallus gallus, défini un nouveau repliement 3D non-répertorié dans la Protein Data Bank. Nous avons étudié plusieurs activités biologiques, en particulier antibactériennes, antiparasitaires et antivirales, et évalué la contribution de chacun des domaines à ses activités (Guyot et al. 2020 à gauche). Enfin, nous avons montré que le gène de cette double-defensine, présent dans tous les génomes d’oiseaux actuellement répertoriés (plus de 300), ne provient pas d’une duplication-fusion (Guyot et al. 2022 à droite).
Les défensines ancestrales d’huitres
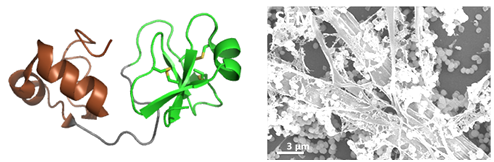
Fig2 : Les big-défensines sont des PAMs à deux domaines, hautement diversifiées chez les mollusques Nous avons synthétisé deux big défensines représentatives de la diversité moléculaire chez l’huître Crassostrea gigas (synthèse peptidique en phase solide et ligation native, coll. Equipe Aucagne), et résolu leur structure 3D (exemple Cg-BigDef1, à gauche). Elles comportent un domaine β-défensine-like (en vert) riche en pont disulfure et un domaine N-terminal hydrophobe, très conservé, structuré en hélice-tour-hélice (en brun). Ces défensines sont actives en milieu salé (400 mM NaCl) contre un large spectre de bactéries. Nous avons montré une synergie d’action entre les domaines, même quand ils ne sont pas liés de façon covalente (Loth et al. 2019) et une complémentarité remarquable de leur spectre antimicrobien (DeSanNicolas et al 2022). On a montré aussi que le domaine hydrophobe est indispensable pour l’activité antimicrobienne en milieu salé, et qu’il gouverne l’auto-association de la molécule entière en formant des « nanonets » pour piéger les bactéries (Loth et al. 2019, à droite).
Les défensines du Manchot Royal
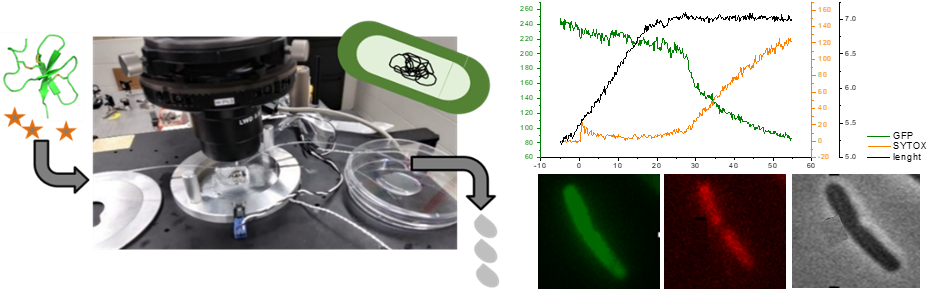
Fig3 : Une première étape essentielle vers l'ingénierie rationnelle de peptides antibactériens hautement efficaces à usage clinique consiste à comprendre en détail le fonctionnement des PAMs optimisés par la nature. Les données concernant les PAMs non lytiques (cad qui ne lysent pas la membrane bactérienne pour détruire la bactérie), tels que les β-défensines, sont rares et restent donc mal comprises. Nous étudions le mécanisme d’action des défensines, sur l’exemple de la défensine AvBD103b du Manchot royal, en réalisant des expériences de microscopie de fluorescence sur bactéries E. coli vivantes qui nous permettent de suivre en temps réel la moindre perméabilisation de leurs membranes interne et externe (Landon et al. 2022).
II. AUTRES PEPTIDES RICHES EN PONTS DISULFURE (TOXINES)
Les toxines riches en pont disulfure ont de fortes identités structurales avec les peptides antimicrobiens décrits ci-dessus. Nous étudions des toxines de diverses origines (plantes, fourmis, araignées, scorpions). Nous travaillons à la compréhension fine des structures (diversités structurales, relations structures-activités, évolution des structures au cours de l’évolution des espèces) pour améliorer la compréhension des mécanismes d’action et participer à la conception des nouveaux composés insecticides de demain. Enfin, nous participons à plusieurs projets sur des toxines photo-activables, ce qui ouvrent de nouvelles voies thérapeutiques.
Toxines insecticides de plantes
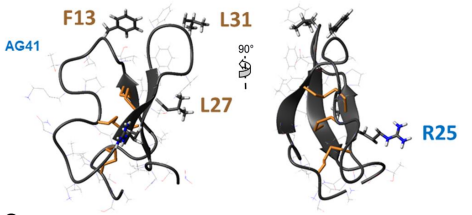
Fig4 : Les biopesticides sont des alternatives efficaces aux insecticides chimiques. Nous étudions le peptide AG41 provenant des racines de la luzerne qui présente une activité insecticide exceptionnelle contre plusieurs ravageurs tels que les pucerons et les charançons. La structure 3D que nous avons déterminée a permis d’établir des relations structure-fonction qui expliquent cette forte activité insecticide. Les résidus clés pour l'entomotoxicité du peptide AG41 sont F13, R25, L27 et L31 (Diya et al 2023).
Toxines insecticides de venins de fourmis
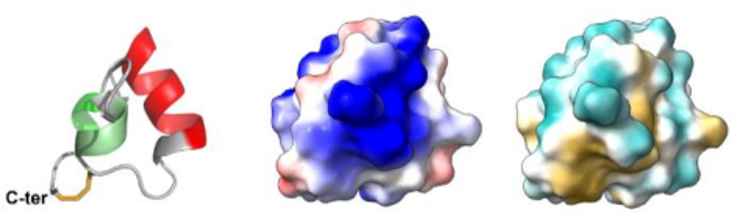
Fig5 : Les venins de fourmis, cocktails de toxines optimisés pour perturber les systèmes physiologiques des insectes proies, constituent un réservoir très prometteur pour la découverte de nouveaux insecticides. Parmi les peptides que nous avons étudiés, nous avons montré que le peptide U11 est l’un des plus paralysants jamais rapportés à partir de venins de fourmis contre les mouches à viande. Le peptide U11 a une structure hélicoïdale compacte unique, stabilisée par un pont disulfure (de gauche à droite la structure 3D du peptide U11, les potentiels électrostatiques à la surface, les potentiels hydrophobes à la surface).Barrassé et al 2023
Toxines de scorpion ou d’araignée photo-activables
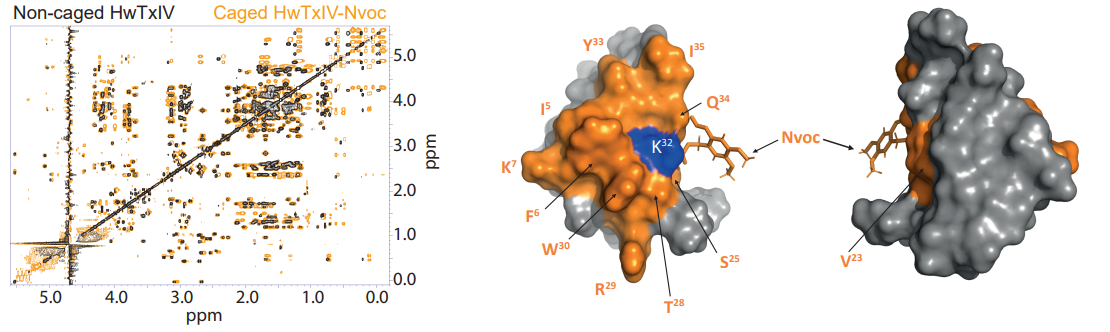
Fig6 : Les nouvelles familles de médicaments photo-activables ouvrent de nouvelles possibilités d'interventions thérapeutiques. Dans ce contexte, nous étudions par exemple des toxines riches en ponts disulfures qui ciblent les canaux sodiques neuronaux (Montnach et al. 2022) ou les canaux cardiaques ERG (Montnach et al. 2023), et qui sont rendues photo-activables par l’ajout de ligands covalents optimisés et clivables par la lumière UV. A gauche exemple de superposition de spectres RMN d’une toxine s’araignée ciblant les canaux ioniques neuronaux, libre ou liée au groupement Nvoc photo-activable. A droite, structure 3D indiquant la zone perturbée par la présence du groupement photo-activable (Montnach et al. 2022)
III. MÉTABOLOMIQUE
Nous développons l’analyse métabolomique par RMN, essentiellement sur des modèles drosophiles de pathologies humaines, telles que les gliomes (Maravat et al. 2021, Bertrand et al. 2023), la maladie de Huntington (Bertrand et al. 2020) et la maladie de Parkinson. D’autres analyses sont aussi mises en œuvre pour étudier différents effets comme celui d’un traitement thérapeutique. L’objectif de ces études est d’identifier les voies métaboliques impactées par les maladies pour mieux les comprendre au niveau moléculaire et permettre l’identification de nouvelles cibles thérapeutiques.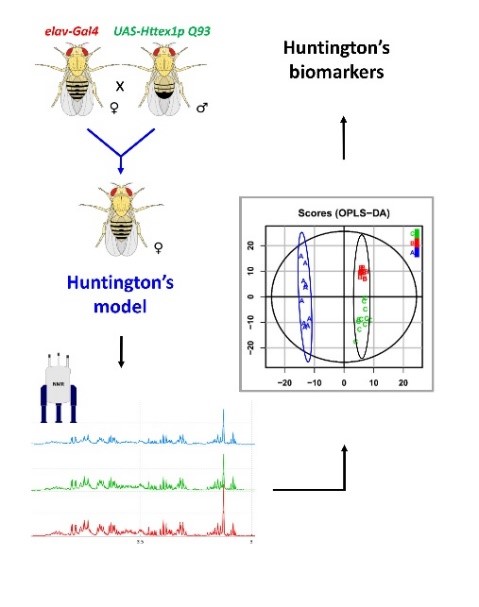 Fig7 : Résumé graphique d’une étude métabolomique sur l’exemple de la maladie de Huntington (Bertrand et al. 2020) : i) Construction du modèle drosophile porteur de la maladie de Huntington, ii) Analyses RMN, iii) Analyses statistiques et iv) Identification des biomarqueurs de la maladie.
Fig7 : Résumé graphique d’une étude métabolomique sur l’exemple de la maladie de Huntington (Bertrand et al. 2020) : i) Construction du modèle drosophile porteur de la maladie de Huntington, ii) Analyses RMN, iii) Analyses statistiques et iv) Identification des biomarqueurs de la maladie.
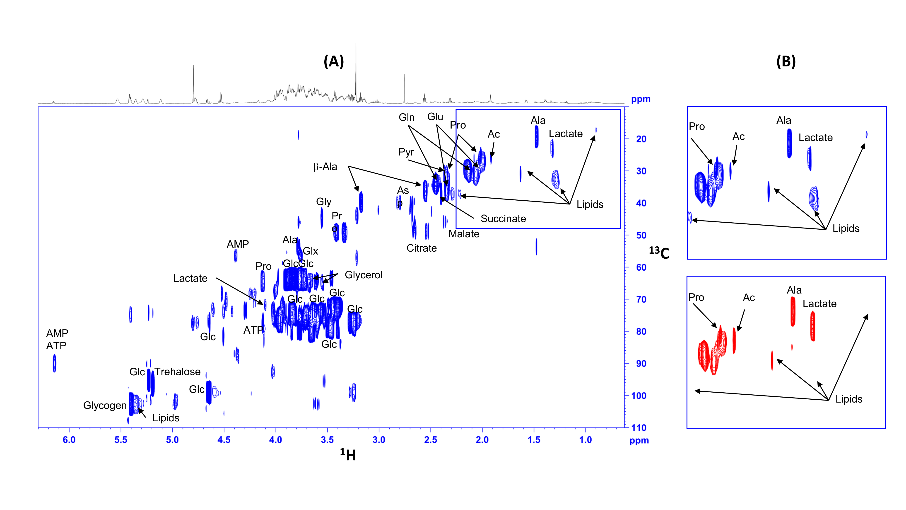 Fig8: Spectres RMN 13C-HSQC d'extraits de drosophiles témoins (bleu) et porteuses de gliomes (rouge), nourries au glucose 13C. Les signaux provenant des lipides (bien visibles sur les agrandissements) sont considérablement réduits chez les mouches porteuses de gliomes, alors que le lactate est augmenté (Bertrand et al. 2023)
Fig8: Spectres RMN 13C-HSQC d'extraits de drosophiles témoins (bleu) et porteuses de gliomes (rouge), nourries au glucose 13C. Les signaux provenant des lipides (bien visibles sur les agrandissements) sont considérablement réduits chez les mouches porteuses de gliomes, alors que le lactate est augmenté (Bertrand et al. 2023)
IV. LA BIOLOGIE STRUCTURALE POUR LE DEVELOPPEMENT DE VACCINS ANTI-SARS-COV-2
Via notre expertise en biologie structurale des protéines, nous avons participé, en collaboration avec l’Institut Finlay des Vaccins de Cuba, au développement des vaccins SOBERANA contre la COVID19, basés sur la protéine recombinante RBD, domaine de fixation de la protéine Spike du SARS-CoV-2 sur le récepteur ACE2.
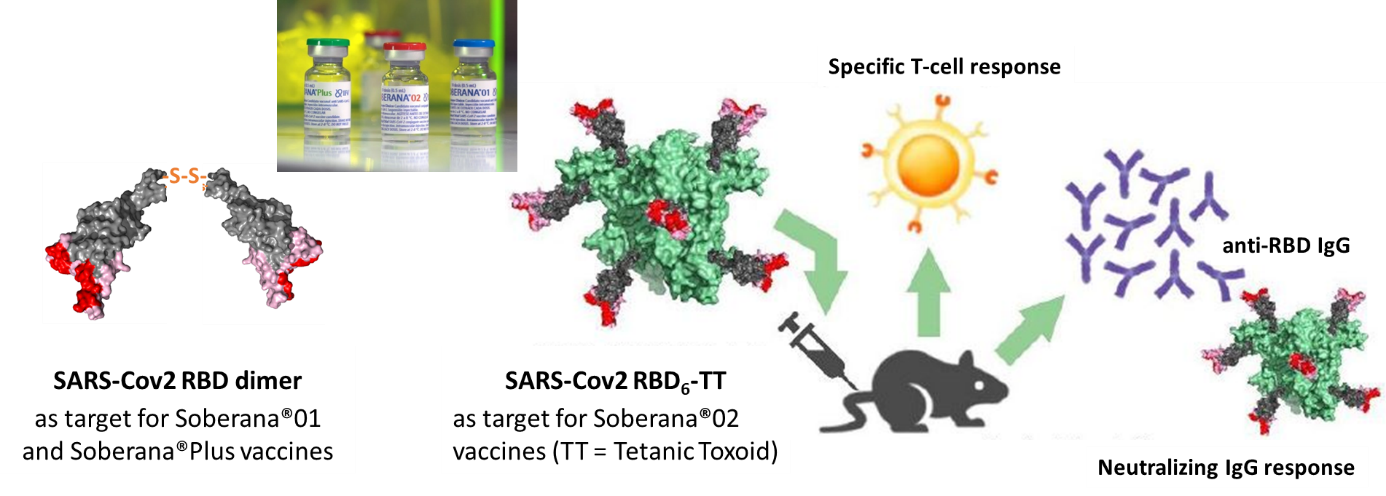
Fig8 : La protéine recombinante RBD entre dans la composition des vaccins SOBERANA (Valdes et al ACS Central Sciences 2021) soit sous forme de dimère (Santana et al 2022), soit conjuguée à l’anatoxine tétanique (Valdes et al ACS ChemBio 2021).
Principales publications (par odre alphabétique du nom du premier auteur) :
- Barasse V, Jouvensal L, Boy G, Billet A, Ascoet S, Lefranc B, Leprince J, Dejean A, Lacotte V, Rahioui I, Sivignon C, Gaget K, Ribeiro Lopes M, Calevro F, Da Silva P, Loth K, Paquet F, Treilhou M, Bonnafe E and Touchard A. Discovery of an Insect Neuroactive Helix Ring Peptide from Ant Venom. Toxins (Basel). 2023;15.
- Bertrand M, Decoville M, Meudal H, Birman S and Landon C. Metabolomic Nuclear Magnetic Resonance Studies at Presymptomatic and Symptomatic Stages of Huntington’s Disease on a Drosophila Model.Journal of Proteome Research
- Bertrand M, Szeremeta F, Hervouet-Coste N, Sarou-Kanian V, Landon C, Morisset-Lopez S and Decoville M. An adult Drosophila glioma model to highlight metabolic dysfunctions and evaluate the role of the serotonin 5‐HT 7 receptor as a potential therapeutic target. FASEB Journal. 2023;37.
- Diya F, Jouvensal L, Rahioui I, Loth K, Sivignon C, Karaki L, Kfoury L, Rizk F and da Silva P. Residues of Legume AG41 Peptide Crucial to Its Bio-Insecticidal Activity. Biomolecules. 2023;13:446.
- Guyot N, Landon C and Monget P. The Two Domains of the Avian Double-β-Defensin AvBD11 Have Different Ancestors, Common with Potential Monodomain Crocodile and Turtle Defensins. Biology. 2022;11.
- Guyot N, Trapp S, Meudal H, Iochmann S, Silvestre A, Jousset G, Labas V, Reverdiau P, Loth K, Herve V, Aucagne V, Delmas AF, Réhault-Godbert S and Landon C. Structure, function, and evolution of Gga -AvBD11, the archetype of the structural avian-double-β-defensin family. Proceedings of the National Academy of Sciences of the United States of America. 2020;117:337-345.
- Landon C, Zhu Y, Mustafi M, Madinier J-B, Lelièvre D, Aucagne V, Delmas A and Weisshaar JC. Real-Time Fluorescence Microscopy on Living E. coli Sheds New Light on the Antibacterial Effects of the King Penguin beta-Defensin AvBD103b. International Journal of Molecular Sciences. 2022;23:2057.
- Loth K, Vergnes A, Barreto C, Voisin SN, Meudal H, Da Silva J, Bressan A, Belmadi N, Bachere E, Aucagne V, Cazevielle C, Marchandin H, Rosa RD, Bulet P, Touqui L, Delmas AF and Destoumieux-Garzon D. The Ancestral N-Terminal Domain of Big Defensins Drives Bacterially Triggered Assembly into Antimicrobial Nanonets. mBio. 2019;10.
- Maravat M, Bertrand M, Landon C, Fayon F, Morisset-Lopez S, Sarou-Kanian V and Decoville M. Complementary Nuclear Magnetic Resonance-Based Metabolomics Approaches for Glioma Biomarker Identification in a Drosophila melanogaster Model. Journal of Proteome Research. 2021;20:3977-3991.
- Montnach J, Blömer L, Ananda, Lopez L, Filipis L, Meudal H, Lafoux A, Nicolas S, Chu D, Caumes C, Béroud R, Jopling C, Bosmans F, Huchet C, Landon C, de Waard M, Blömer LA and Canepari M. In vivo spatiotemporal control of voltage-gated ion channels by using photoactivatable peptidic toxins. Nature Communications. 2022;13:417.
- Montnach J, Millet H, Persello A, Meudal H, de Waard S, Mesrica P, Ribeiro B, Richard J, Hivonnait A, Tessier A, Lauzier B, Charpentier F, Mangoni M, E, Landon C, Jopling C and de Waard M. Optical Control of Cardiac Rhythm by In Vivo Photoactivation of an ERG Channel Peptide Inhibitor. Circulation Research. 2023;133:535-538.
- Nicolas NdS, Asokan A, Rosa R, Voisin S, Travers M-A, Rocha G, Dantan L, Dorant Y, Mitta G, Petton B, Charrière G, Escoubas J-M, Boulo V, Pouzadoux J, Meudal H, Loth K, Aucagne V, Delmas A, Bulet P, Montagnani C and Destoumieux-Garzón D. Functional Diversification of Oyster Big Defensins Generates Antimicrobial Specificity and Synergy against Members of the Microbiota. Marine drugs. 2022;20:745.
- Santana D, Perez Nicado R, Climent Y, Rodríguez Noda LMM, Sánchez Ramírez B, Perez-Rodriguez S, Rodriguez M, Labrada Regalado C, Hernandez T, Diaz M, Orosa I, Ramirez U, Oliva R, Garrido R, Cardoso F, Landys M, Martinez R, Gonzalez H, Hernandez T, Ochoa-Azze R, Perez J, Enriquez J, Gonzalez N, Infante Y, Espinosa LA, Ramos Y, Gonzalez LJ, Valenzuela C, Casadesus AV, Fernandez B, Rojas G, Pérez-Massón B, Tundidor Y, Bermudez E, Plasencia C, Boggiano T, Ojito E, Chiodo F, Fernandez S, Paquet F, Fang C, Chen G-W, Rivera D, Valdes Y, Garcia-Rivera D and Verez Bencomo V. A COVID-19 vaccine candidate composed of SARS-CoV-2 RBD dimer and Neisseria meningitidis outer membrane vesicles. RSC Chemical Biology. 2022;3:242-249.
- Valdes-Balbin Y, Santana-Mederos D, Quintero L, Fernandez S, Rodriguez L, Sanchez Ramirez B, Perez-Nicado R, Acosta C, Mendez Y, Ricardo MG, Hernandez T, Bergado G, Pi F, Valdes A, Carmenate T, Ramirez U, Oliva R, Soubal JP, Garrido R, Cardoso F, Landys M, Gonzalez H, Farinas M, Enriquez J, Noa E, Suarez A, Fang C, Espinosa LA, Ramos Y, Gonzalez LJ, Climent Y, Rojas G, Relova-Hernandez E, Cabrera Infante Y, Losada SL, Boggiano T, Ojito E, Leon K, Chiodo F, Paquet F, Chen GW, Rivera DG, Garcia-Rivera D and Verez Bencomo V. SARS-CoV-2 RBD-Tetanus Toxoid Conjugate Vaccine Induces a Strong Neutralizing Immunity in Preclinical Studies. ACS Chem Biol. 2021;16:1223-1233.
