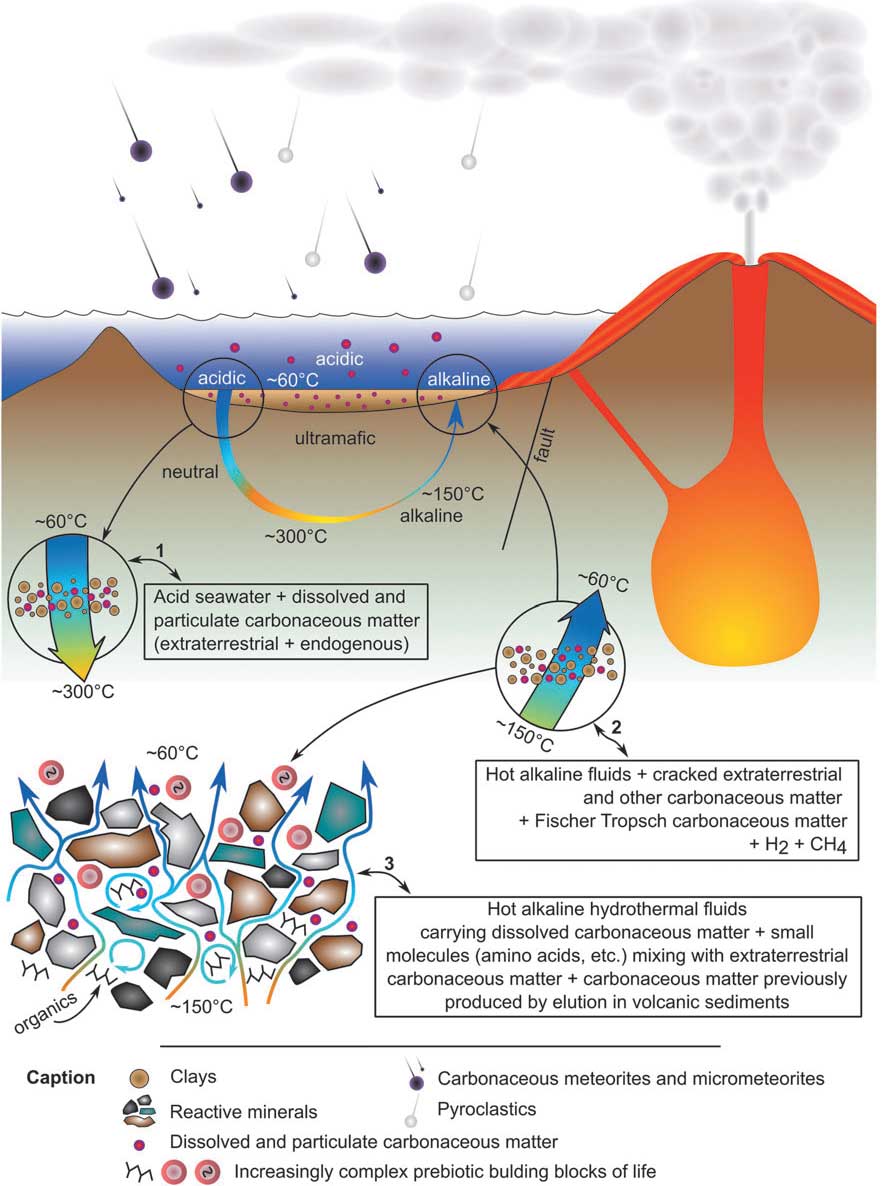
L'origine de la vie est l'une des questions les plus fondamentales de l'humanité. Elle est abordée par un large éventail de chercheurs de différents domaines, avec des approches et des idées différentes quant à la façon dont elle est apparue. Cependant, ce qui manque aux expériences de chimie prébiotique, ce sont des informations précises sur l'environnement et les conditions régnant sur la Terre primitive à l'ère Hadéenne (4,5-4,0 Ga). En particulier, il y a un manque de connaissance sur les ingrédients inorganiques qui étaient disponibles, la stabilité et la longévité des divers environnements suggérés comme lieux d'émergence de la vie, ainsi que la cinétique et la fréquence des étapes prébiotiques ayant conduit aux premiers systèmes vivants.
Cette contribution passe en revue notre compréhension actuelle de la géologie de la Terre primitive à l'époque où la vie a émergé. Après avoir décrit le scénario géologique, nous évoquons les questions encore ouvertes sur l'origine de la vie : la vie a-t-elle commencé organiquement ou sous forme minéralogique ? Si organiquement, quelle était l'origine des constituants organiques de la vie ? Qu'est-ce qui est venu en premier, le métabolisme ou la réplication ? Quelle a été l'échelle de temps pour l'émergence de la vie? Nous concluons que la voie à suivre pour la chimie prébiotique est une approche fusionnant géologie et chimie, c'est-à-dire des cycles de réactions loin de l'équilibre se produisant de manière répétée et itérative sur des surfaces minérales dans des conditions hydrothermales.
Setting the geological scene for the origin of life and continuing open questions about its emergence
Frances Westall1, André Brack, Alberto G. Fairén and Mitchell D. Schulte
Frontiers in Astronomy and Space Sciences - 05 January 2023 - Volume 9 - doi : 10.3389/fspas.2022.1095701 9:1095701