Cette équipe étudie les relations structure-fonction des systèmes biologiques contenant de l’ARN. Expert des protéines de remodelage de l’ARN, l'équipe développe des approches biochimiques et bioinformatiques sophistiquées pour décrypter les mécanismes moléculaires de ces facteurs essentiels.
Descriptif du travail de recherche
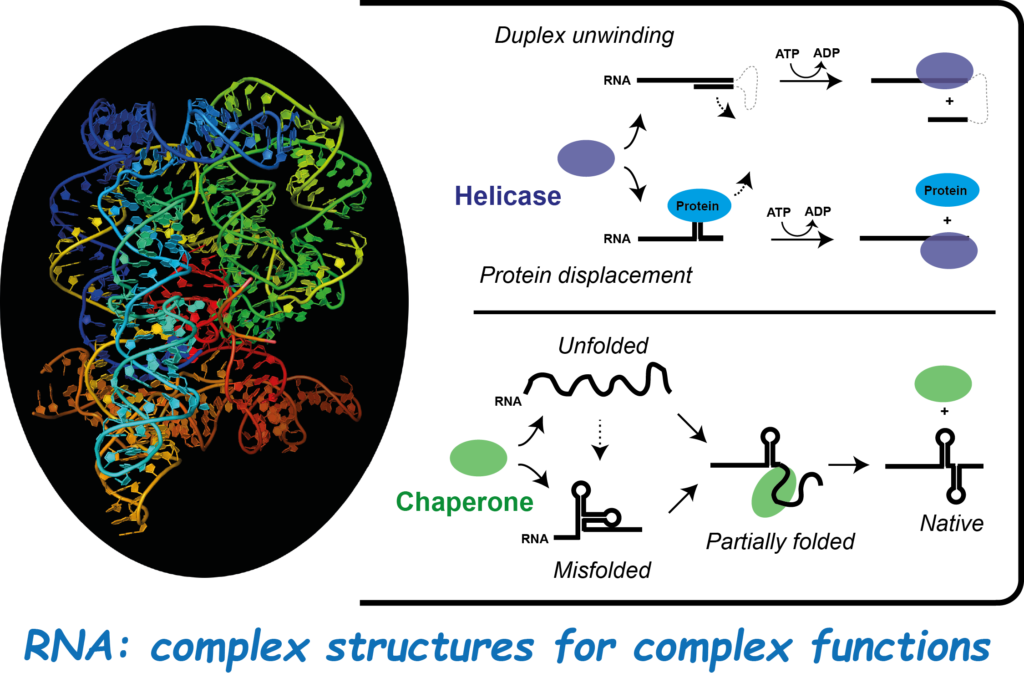
L'équipe est particulièrement intéressé par les mécanismes de remodelage de la structure des ARN et des complexes ribonucléoprotéiques (RNP). Cette structure, généralement dynamique, peut évoluer au cours de la vie cellulaire et gouverne le rôle et le devenir des ARN. Deux familles principales de protéines ubiquitaires - ARN chaperones et ARN hélicases - assurent la structuration et le remodelage des ARN et des complexes RNP. Ces protéines sont impliquées dans diverses pathologies dont certains cancers. Nous étudions comment ARN hélicases et ARN chaperones fonctionnent et coopèrent pour moduler les fonctions biologiques des ARN.
La fixation des ARN chaperones à l’ARN provoque généralement la destruction de structures secondaires et la formation de motifs ou assemblages structuraux alternatifs. Les ARN hélicases, elles, utilisent l’énergie dérivée de leur activité NTPase pour détruire des régions ARN en double-brin, des hétéroduplexes ARN-ADN et/ou des complexes RNP. Ces propriétés résultent parfois d’une activité de translocation directionnelle le long de l’ARN.
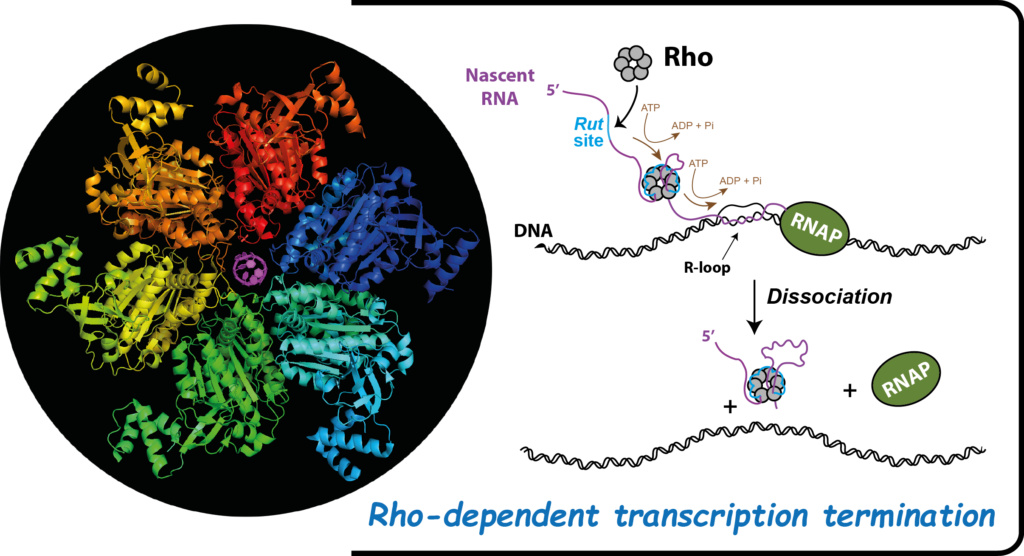 Pour étudier les mécanismes de remodelage de l’ARN, nous utilisons des approches complémentaires de biologie, de biochimie et de biophysique. Nous concentrons nos efforts sur l’étude d’un système modèle complexe au cœur duquel se trouve Rho, un moteur moléculaire ‘six-cylindres’ à activité ARN hélicase impliqué dans la terminaison de la transcription et le maintien de l’intégrité génomique chez les bactéries.
Pour étudier les mécanismes de remodelage de l’ARN, nous utilisons des approches complémentaires de biologie, de biochimie et de biophysique. Nous concentrons nos efforts sur l’étude d’un système modèle complexe au cœur duquel se trouve Rho, un moteur moléculaire ‘six-cylindres’ à activité ARN hélicase impliqué dans la terminaison de la transcription et le maintien de l’intégrité génomique chez les bactéries.
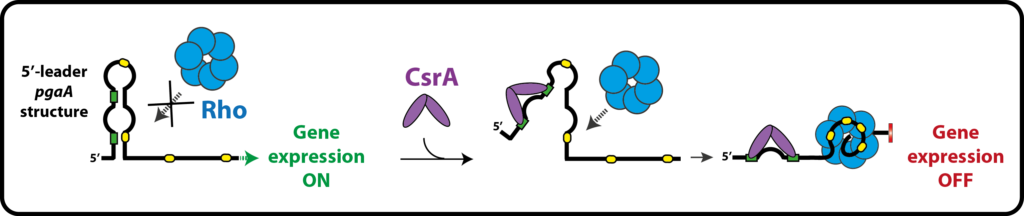
Nous étudions les mécanismes moléculaires gouvernant l’activité de Rho, son rôle dans les mécanismes de régulation génique impliquant ARN non-codants et ARN chaperones (Hfq, CsrA), son importance pour la pathogénicité et l’adaptabilité bactérienne, la conservation de ses fonctions et mode(s) d’action au sein de la biodiversité, etc.
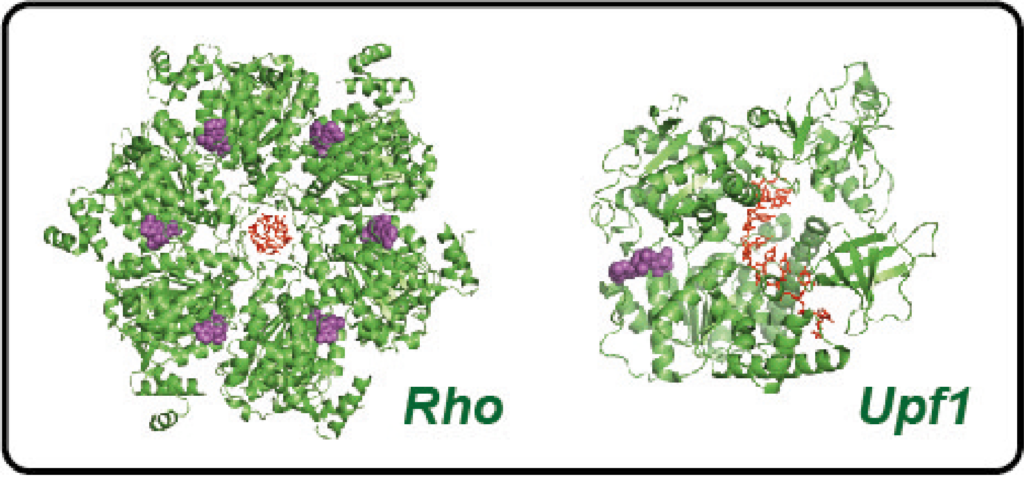 Nous étudions également d’autres ARN hélicases, en particulier celles qui, a priori, remodèlent l’ARN via des mécanismes distincts de ceux de Rho. De cette façon, nous espérons appréhender dans leur ensemble les mécanismes fondamentaux qui gouvernent ces enzymes, déterminer les points communs ou les différences qui les caractérisent au niveau moléculaire et identifier les facteurs qui contribuent à leur régulation.
Nous étudions également d’autres ARN hélicases, en particulier celles qui, a priori, remodèlent l’ARN via des mécanismes distincts de ceux de Rho. De cette façon, nous espérons appréhender dans leur ensemble les mécanismes fondamentaux qui gouvernent ces enzymes, déterminer les points communs ou les différences qui les caractérisent au niveau moléculaire et identifier les facteurs qui contribuent à leur régulation.
L’objectif, à terme, est d’exploiter cette information à des fins de ciblage thérapeutique ou de conception de bio-senseurs et de circuits géniques inductibles intéressants pour la biologie synthétique
Principales Publications :
- Cédric Nadiras, Eric Eveno, Annie Schwartz, Nara Figueroa-Bossi, Marc Boudvillain.
A multivariate prediction model for Rho-dependent termination of transcription.
Nucleic Acids Research (2018) 46, 8245-8260 - Odil Porrua, Marc Boudvillain, Domenico Libri.
Transcription Termination: Variations on Common Themes.
Trends in Genetics (2016) 32, 508-522. - Emilie Soares, Annie Schwartz, Marcello Nollmann, Emmanuel Margeat, Marc Boudvillain.
The RNA-mediated, asymmetric ring regulatory mechanism of the transcription termination Rho helicase decrypted by time-resolved nucleotide analog interference probing (trNAIP).
Nucleic Acids Research (2014) 9270-9284. - Nara Figueroa-Bossi, Annie Schwartz, Benoit Gillemardet, François D’Heygère, Lionello Bossi, Marc Boudvillain
RNA remodeling by bacterial global regulator CsrA promotes Rho-dependent transcription termination
Genes & Development (2014) 28, 1239-51. - Rachid Menouni, Stéphanie Champ, Léon Espinosa, Marc Boudvillain, Mireille Ansaldi
Transcription termination controls prophage maintenance in Escherichia coli genomes
Proc. Natl. Acad. Sci USA (2013) 110, 14414-14419.