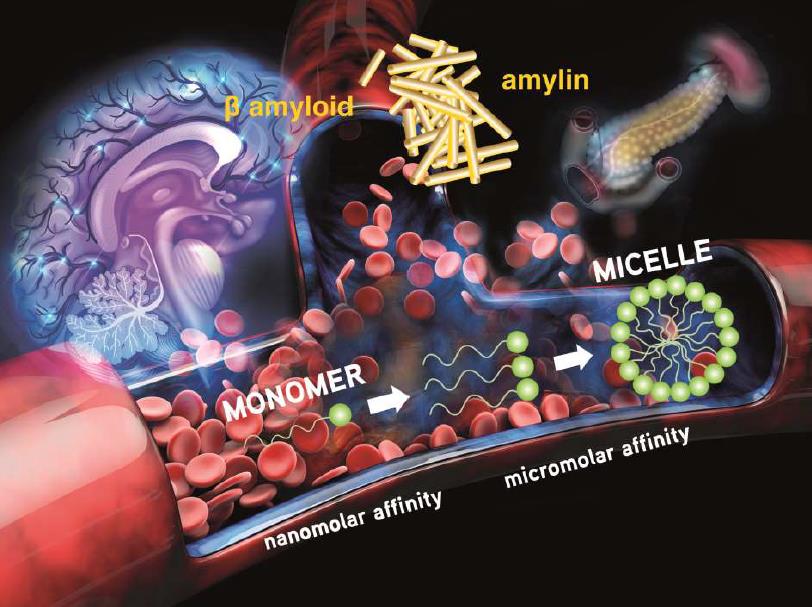
De nombreux complexes métalliques qui ciblent les peptides amyloïdes ont été proposés en tant qu’agents d’imagerie pour la détection des pathologies amyloïdogéniques. Par exemple, des chélates de gadolinium peuvent être utilisés en IRM, ou des radiocomplexes (64Cu, 99mTc, etc) en imagerie nucléaire. D’autres complexes, capables d’empêcher la formation des dépôts amyloïdes, sont proposés pour la thérapie.
Toutes ces molécules sont amphiphiles, comprenant une partie hydrophile (qui contient le métal) et une partie hydrophobe (capable de reconnaitre ces dépôts). Grâce à cette structure, elles forment des agrégats micellaires en solution.
Les équipes du CBM et du LCC (Toulouse) ont découvert que cette propriété de micellisation a des conséquences drastiques et imprévues sur leur capacité de reconnaissance des peptides amyloïdes ciblés, mais également sur leur biodistribution in vivo.
Ils ont conçu une nouvelle sonde avec une affinité nanomolaire aux peptides amyloïdes Aβ et amyline, biomarqueurs respectives de la maladie d’Alzheimer et du diabète.
Cette affinité, exceptionnelle pour un complexe métallique, est atteinte uniquement si les molécules sont sous forme « individuelle ». Une fois sous forme micellaire, l’affinité chute de trois ordres de grandeur.
Ces résultats ont des conséquences directes sur la conception des sondes d’imagerie et thérapie des maladies amyloïdogéniques.
Voir l'article