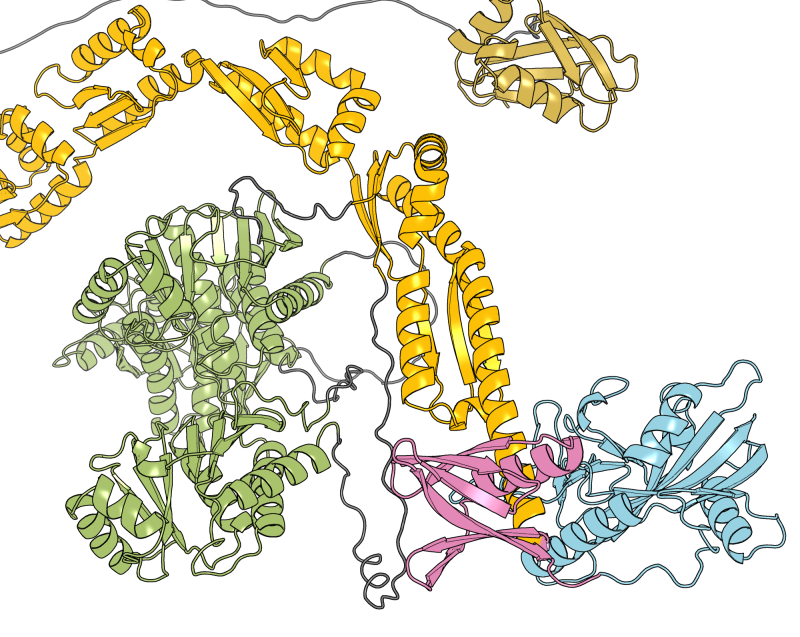
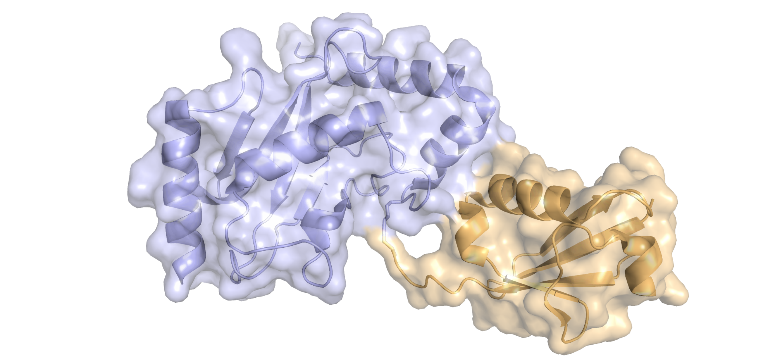
Parmi les principaux éléments fonctionnels des cellules vivantes figurent les protéines, de petites « machines moléculaires » produites par la cellule en fonction des informations codées dans les gènes.
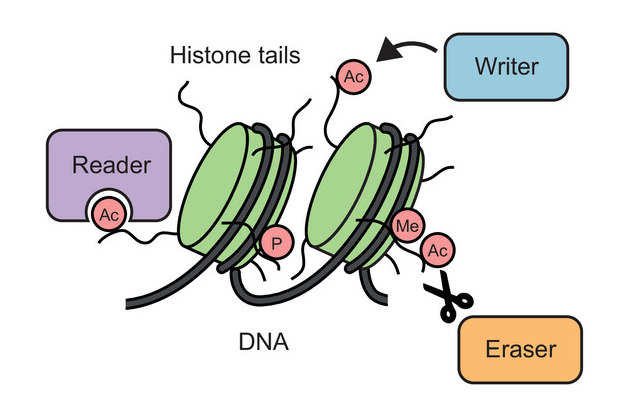
Chaque protéine est caractérisée par une composition chimique qui définit sa structure et sa fonction. Dans certaines circonstances, la composition chimique d'une protéine peut être modifiée au cours d'un processus enzymatique appelé modification post-traductionnelle des protéines (PTM), par lequel des groupes chimiques supplémentaires sont attachés de manière covalente à la protéine. Les PTM sont utilisés par la cellule comme mécanisme de régulation pour contrôler la fonction des protéines. L’ajout de nouveaux groupes chimiques, qui peuvent se présenter sous différentes formes et tailles, allant des petits groupes aux petites protéines en passant par les sucres et les lipides, modifie la structure et les interactions d’une protéine et peut avoir un impact sur presque tous les aspects de sa fonction.
Marcin Suskiewicz, biologiste structuraliste et biochimiste du CBM, a consacré de nombreuses années à l'étude de différents types de PTM et supervise actuellement un projet consacré à un type particulier de PTM : la SUMOylation des protéines.
Dans l'article de synthèse publié dans la revue BioEssays, il passe en revue l’historique de la recherche sur les PTM ainsi que diverses facettes de ce phénomène, notamment les principes chimiques sous-jacents, les mécanismes moléculaires et l’évolution. La revue combine une introduction au domaine avec un aperçu de la littérature récente et de nouvelles idées et hypothèses.
Références de l'article :
The logic of protein post-translational modifications (PTMs): Chemistry, mechanisms and evolution of protein regulation through covalent attachments
Marcin Suskiewicz
BioEssays
First published:21 January 2024
https://doi-org.insb.bib.cnrs.fr/10.1002/bies.202300178